GO/Pathway 富集分析
GO 功能分析
Gene Ontology(简称GO)是一个国际标准化的转录本功能分类体系,提供了一套动态更新的标准词汇表(controlled vocabulary)来全面描述生物体中转录本和转录本产物的属性。GO总共有三个ontology(本体),分别描述转录本的分子功能(molecular function)、细胞组分(cellular component)、参与的生物过程(biological process)。GO的基本单位是term(词条、节点),每个term都对应一个属性。 GO功能分析一方面给出差异表达转录本的GO功能分类注释;另一方面给出差异表达转录本的GO功能显著性富集分析。 首先,我们将差异表达转录本向GO数据库(http://www.geneontology.org/)的各term映射,并计算每个term的转录本数,从而得到具有某个GO功能的转录本列表及转录本数目统计。然后应用超几何检验,找出与整个转录本组背景相比,在差异表达转录本中显著富集的GO条目,该假设检验的p-value计算公式为:
其中,N为所有Unigene中具有GO注释的转录本数目;n为N中差异表达转录本的数目;M为所有Unigene中注释为某特定GO term的转录本数目;m为注释为某特定GO term的差异表达转录本数目。计算得到的pvalue通过FDR校正之后,以Q-value≤0.05为阈值,满足此条件的GO term定义为在差异表达转录本中显著富集的GO term。通过GO功能显著性富集分析能确定差异表达转录本行使的主要生物学功能。
如下表为GO功能富集分析结果,第一列为GO term的ID,点击GO ID,可显示这个GO term包含的所有基因,再点击这个GO ID,就可以链接到http://amigo.geneontology.org 官网,可以查看GO的具体信息;第二列为GO term的功能描述;第三列为差异表达基因总数(表头数字)、注释到某个GO term的差异基因数以及占总差异基因数目的百分比;第四列为所有基因总数(表头数字)、注释到某个GO term的基因数以及占总基因数目的百分比;第五列为P value;第六列为多重检验校正后的p.adjust(即qvalue)。注释得到的GO term按照pvalue和qvalue从小到大排列,其中小于差异标准阈值0.05的pvalue和qvalue标红显示。例如,membrane part为在差异表达基因中最显著富集的一个GO term。
GO 分类柱状图说明
同时对差异基因按上下调进行GO term分类统计。如图所示,横坐标代表GO三个 ontology:分子功能(molecular function)、细胞组分(cellular component)、参与的生物过程(biological process)的更细一级分类;由于一个基因常常会有多个不同功能,因此同一个基因会在不同分类条目下出现,每个柱状图统计相互独立;纵坐标代表每个分类条目所对应的基因数目。
GO 富集分析热图说明
pv(qv).go.pdf/png:GO富集分析P值(Q值)热图,过滤标准为至少一个比较组小于等于0.05。左边颜色条代表三个不同的GO Ontology。每一行代表一个GO Term,每一列代表一个比较组。
rf.go.pdf/png:GO富集分析rich factor热图,过滤标准为至少一个比较组大于等于0.05,左边颜色条代表三个不同的GO Ontology。每一行代表一个GO Term,每一列代表一个比较组。
GO 有向无环图说明
从整体上来看,GO注释系统是一个有向无环图(Directed Acyclic Graphs),GO各term之间的关系是单向的,GO term之间的分类关系有三种:“is a”、“part of”和 “regulates”。流程结果会分别给出GO三个ontology(细胞组分、分子功能、生物过程)的有向无环图。
在这个图中,越接近根结点的GO term越概括,往下分支的GO term为注释到更细层级的term。每个结点里,第一行为GO号;第二行为GO功能描述;第三行为P value,当大于0.05时显示为NA;第四行前面的数字为差异转录本注释到该term的转录本数目,后面的数字为所有转录本注释到该term的转录本数目。
形状的含义:程序默认把显著性最高的前10个GO term设置为方形,其他的GO term为圆形。
颜色的含义:颜色越深,代表该GO term越显著。显著程度从低到高所对应的颜色由浅到深分别为:白色——浅黄——深黄——红色。
KEGG Pathway分析
在生物体内,不同转录本相互协调行使其生物学,基于Pathway的分析有助于更进一步了解转录本的生物学功能。KEGG是有关Pathway的主要公共数据库。 Pathway显著性富集分析以KEGG Pathway为单位,应用超几何检验,找出与整个转录本组背景相比,在差异表达转录本中显著性富集的Pathway。
该假设检验的p-value计算公式同GO功能显著性富集分析的相似,计算公式如下:
其中,N为所有Unigene中具有Pathway注释的转录本数目;n为N中差异表达转录本的数目;M为所有Unigene中注释为某特定Pathway的转录本数目;m为注释为某特定Pathway的差异表达转录本数目。 经过多重检验校正后,选择Qvalue≤0.05的Pathway定义为在差异表达转录本中显著富集的Pathway。Q-value和FDR类似,都是对p-value的一种校正。某一个假设检验的Q-value就是FDR的最小值,在这些FDR下该假设检验可以被认为是显著的。注释得到的Pathway按照pvalue和qvalue从小到大排列,其中小于差异标准阈值0.05的pvalue和qvalue标红显示。
通过Pathway显著性富集能确定差异表达转录本参与的最主要生化代谢途径和信号转导途径。
下表为pathway功能富集分析结果,各列含义与GO功能富集分析结果类似。
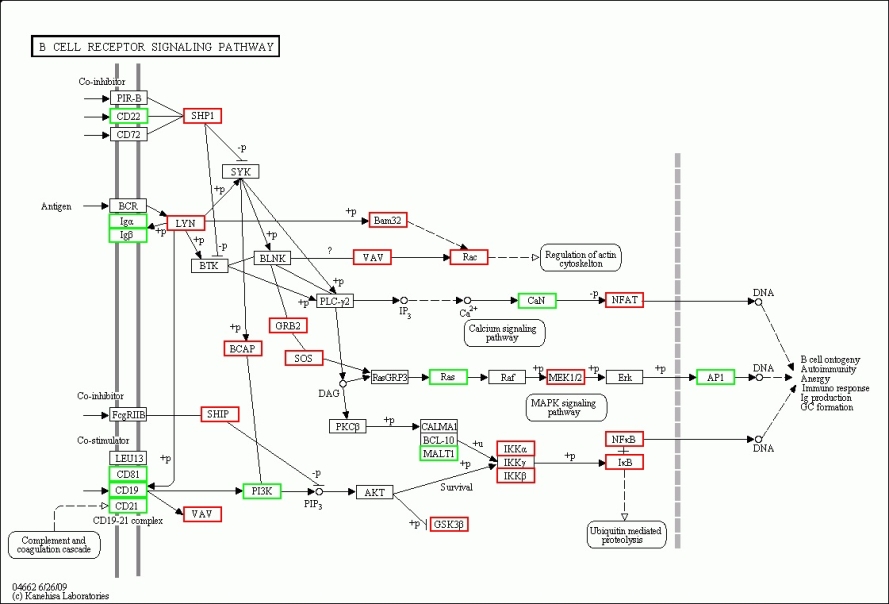
差异表达转录本的Pathway显著性富集分析不但得到最有意义的Pathway列表,点击其中的Pathway链接还将得到KEGG数据库中Pathway的详细信息,如点击表8第一列第三行的B cell receptor signaling pathway,可以看到如图4所示的详细信息,上调转录本所在位置用红色标记,下调转录本所在位置用绿色标记。
KEGG 富集气泡图说明
如图所示,RichFactor指差异表达的转录本中位于该pathway条目的转录本数目与所有转录本中位于该pathway条目的转录本总数的比值,RichFactor越大,表示富集的程度越高。QValue是做过多重假设检验校正之后的PValue,取值范围为0到1,越接近于零,表示富集越显著。该图是用Qvalue从小到大排序前20的pathway来作图的。
KEGG 富集分析热图说明
pv(qv).kegg.pdf/png:KO富集分析P值(Q值)热图,过滤标准为至少一个比较组小于等于0.05。左边颜色条代表KEGG B Class Pathway注释。每一行代表一个KEGG Pathway注释,每一列代表一个比较组。
rf.kegg.pdf/png:KO富集分析rich factor热图,过滤标准为至少一个比较组大于等于0.05,左边颜色条代表KEGG B Class Pathway注释。每一行代表一个KEGG Pathway注释,每一列代表一个比较组。