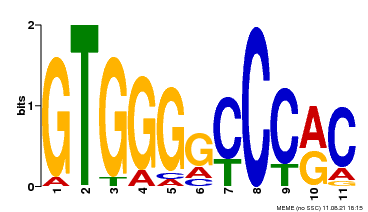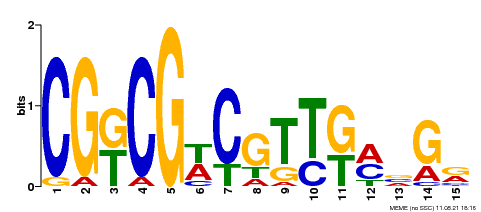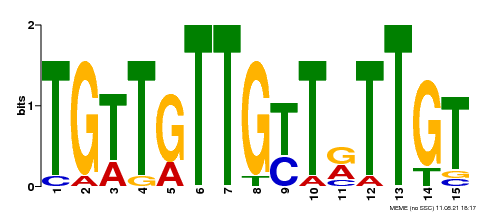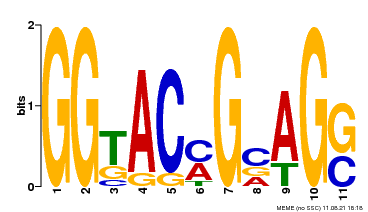1 项目概述
| 项目编号 | GDE21030165-1_std_1 |
| 项目名称 | Capsicum annuum ------ 2 DAP-seq |
| 参考基因组 | Annuum.v1.6 |
| 样品名称 | HY5-IP ¦ HY5-input |
2 项目介绍
2.1 建库流程
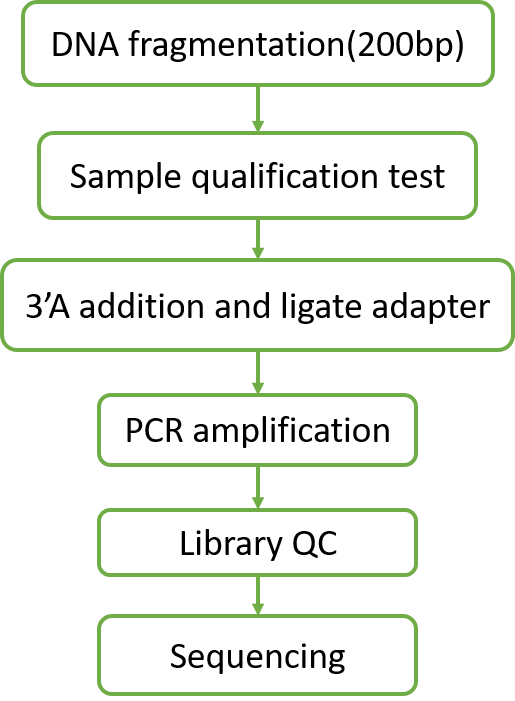 |
| Fig 2-1-1 DAP-seq实验建库流程图 |
2.2 信息分析流程
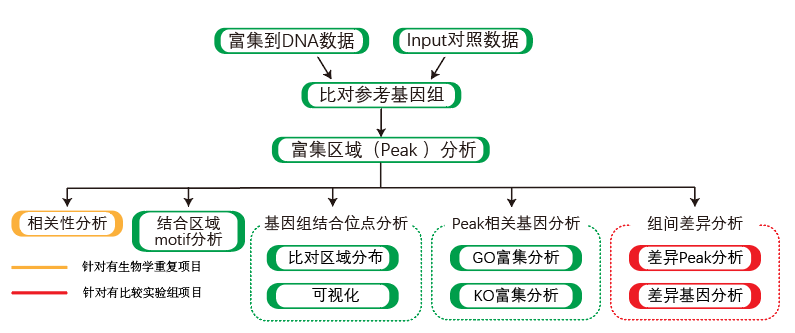 |
| Fig 2-2-1 DAP-seq分析流程图 |
3 数据分析与质控
3.1 过滤信息统计
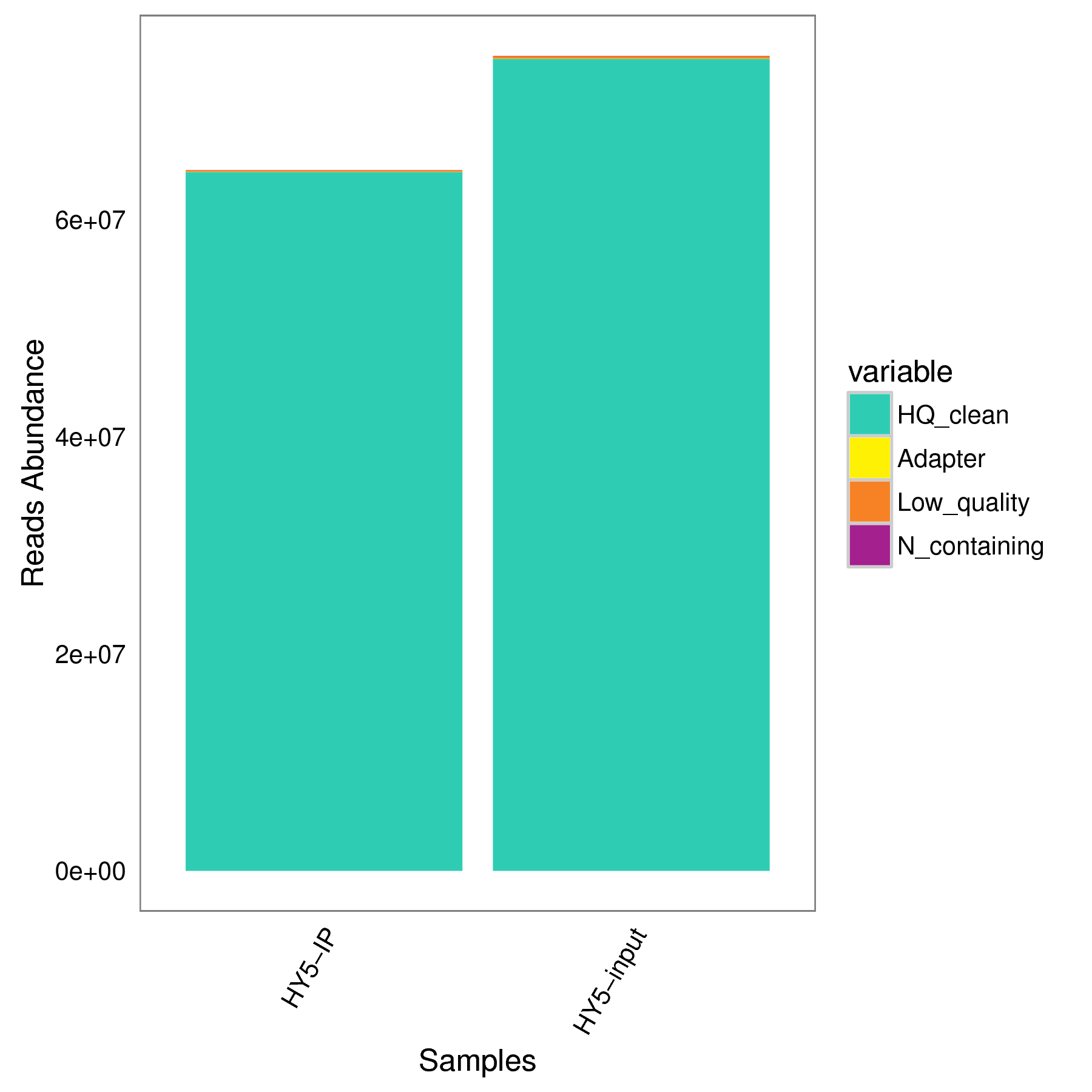 |
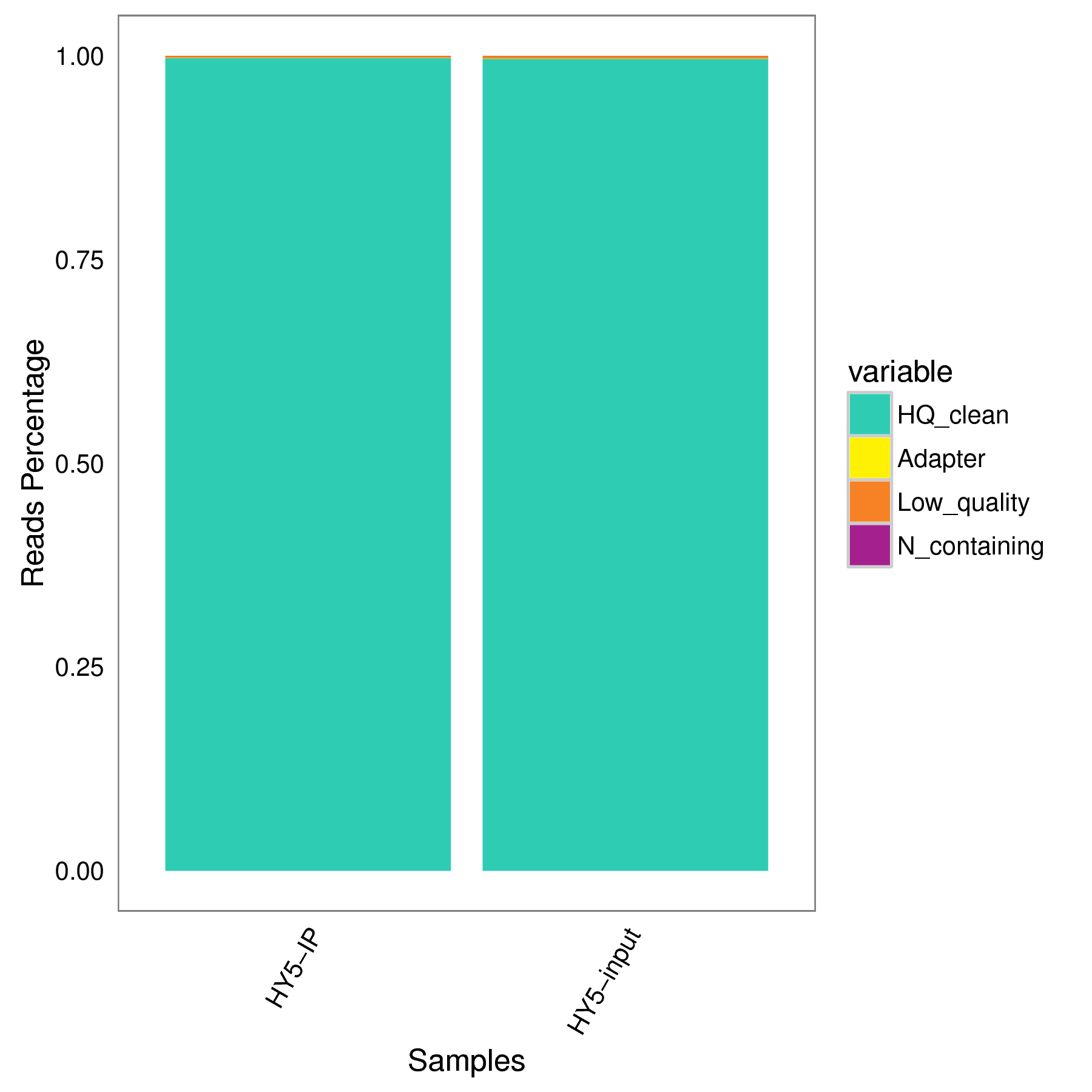 |
|
| Fig 3-1-1 样本过滤分析频数图 | Fig 3-1-2 样本过滤分析比例图 | |
3.2 碱基组成与质量分析
4 比对分析
4.1 比对基因组统计
4.2 基因组测序深度累积分布
4.3 基因组测序深度分布
4.4 测序饱和度分析
- HY5-IP
- HY5-input
Fig 4-4-1 测序饱和度分布
4.5 Reads在染色体上的分布
5 Peak分析
5.1 PeakCalling
| SampleId | PeakNumber | TotalLength | AverageLength | TotalPileup | AveragePileup | GenomeRatio(%) | FRiP(%) |
|---|---|---|---|---|---|---|---|
| HY5-IP | 23561 | 5795060 | 245 | 230891 | 9 | 0.19% | 3.09% |
- HY5-IP peaks结果信息统计表: /3.peak_analysis/HY5-IP_peaks.xls
5.2 Peak长度分布
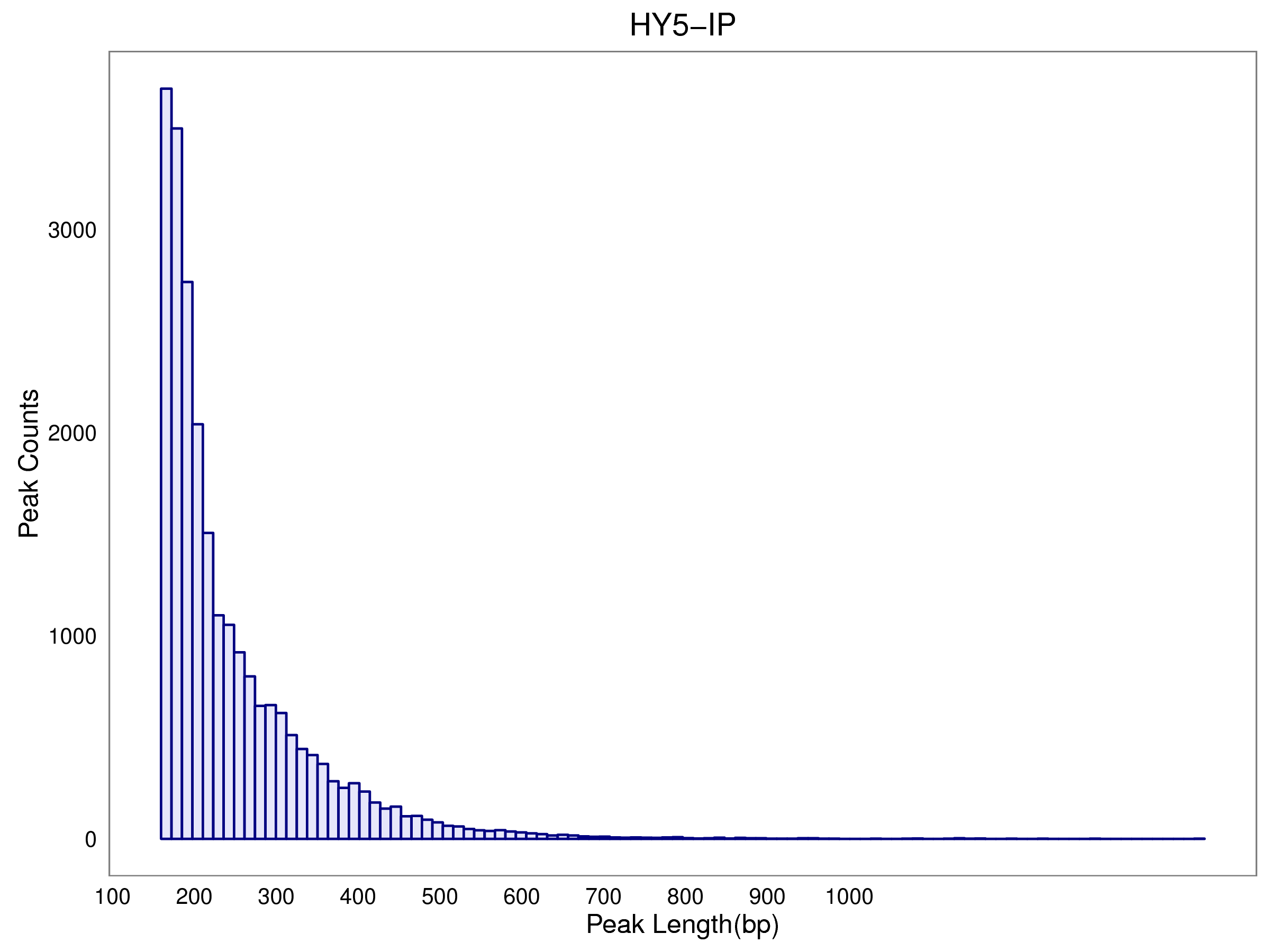 |
Fig 5-2-1 HY5-IP |
5.3 Peak深度分布
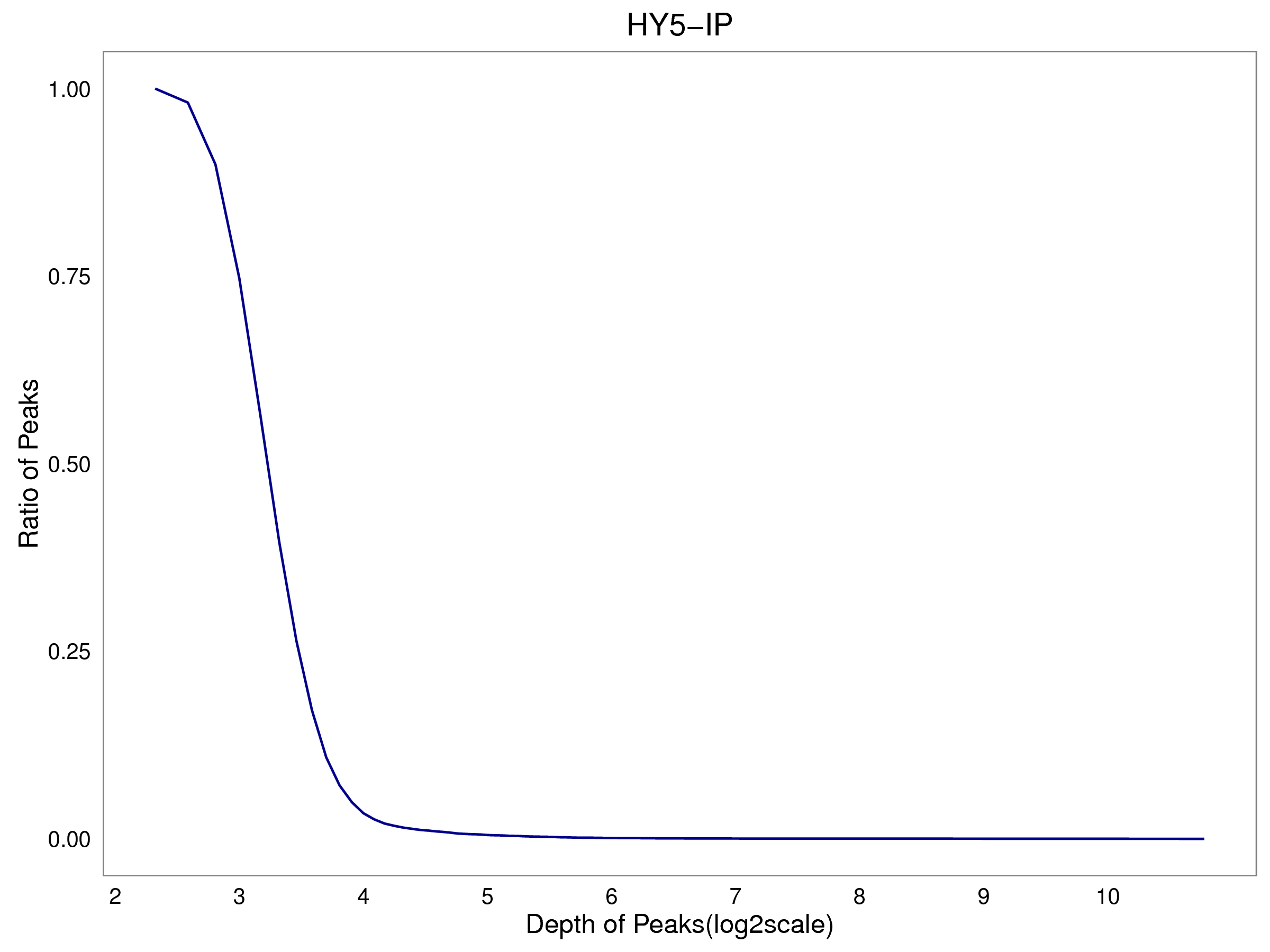 |
Fig 5-3-1 HY5-IP |
5.4 Peak富集倍数分布
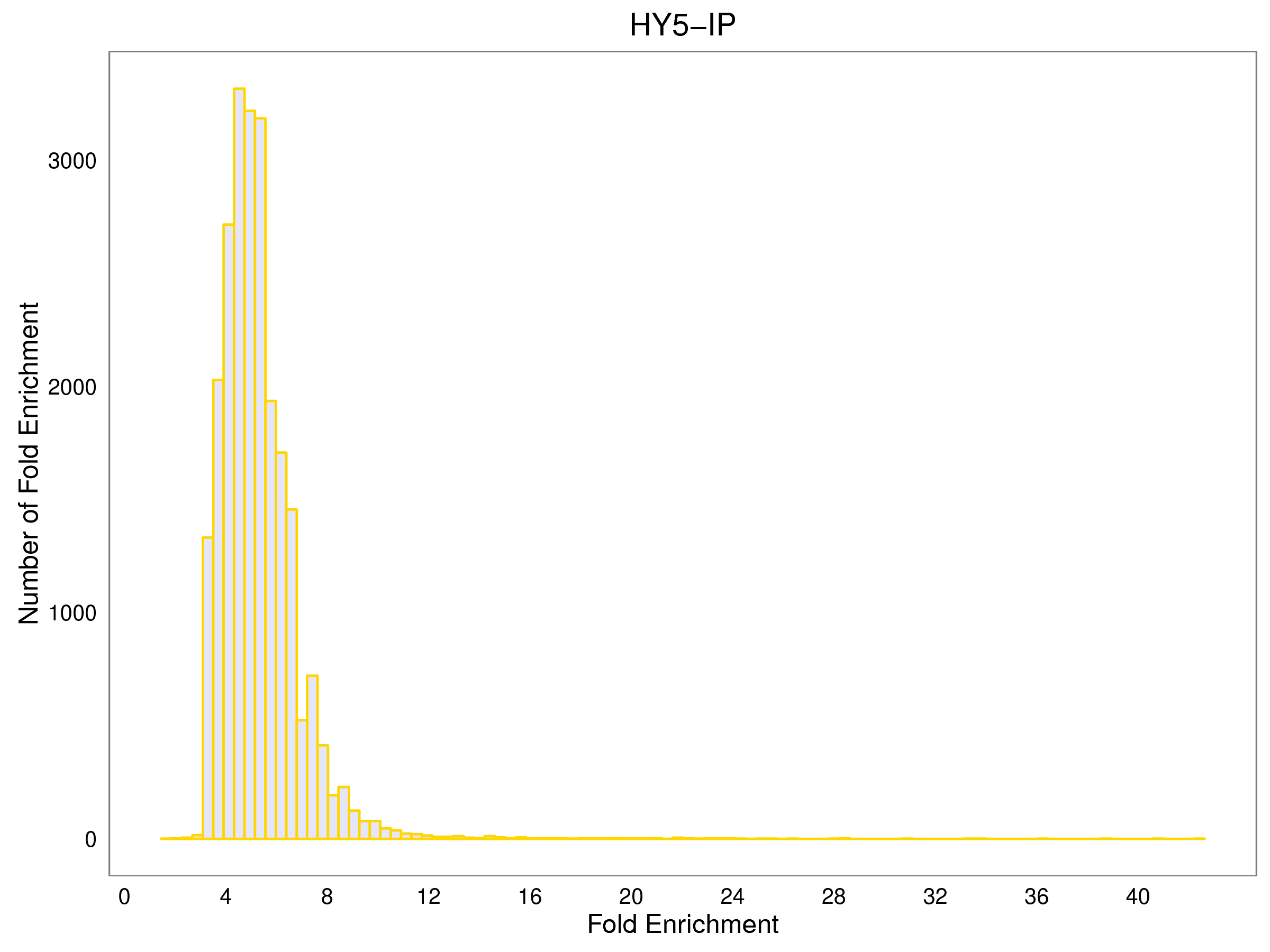 |
Fig 5-4-1 HY5-IP |
5.5 Peak置信程度分布
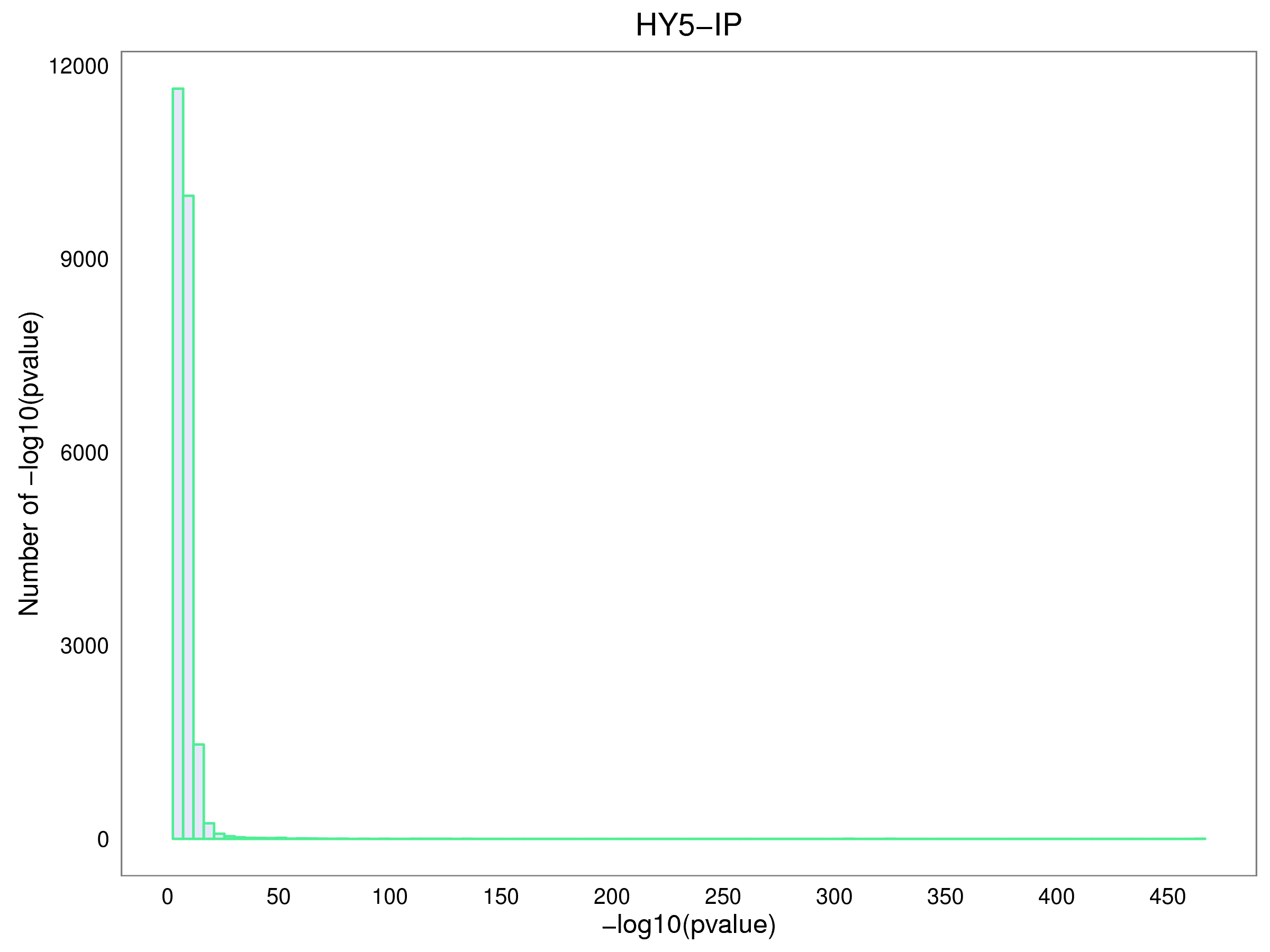 |
Fig 5-5-1 HY5-IP |
6 Peak注释
6.1 Peak相关基因分析
- HY5-IP peak相关基因注释表: /4.peak_annotation/HY5-IP.final.anno.xls
6.2 Peak在基因功能元件上的分布
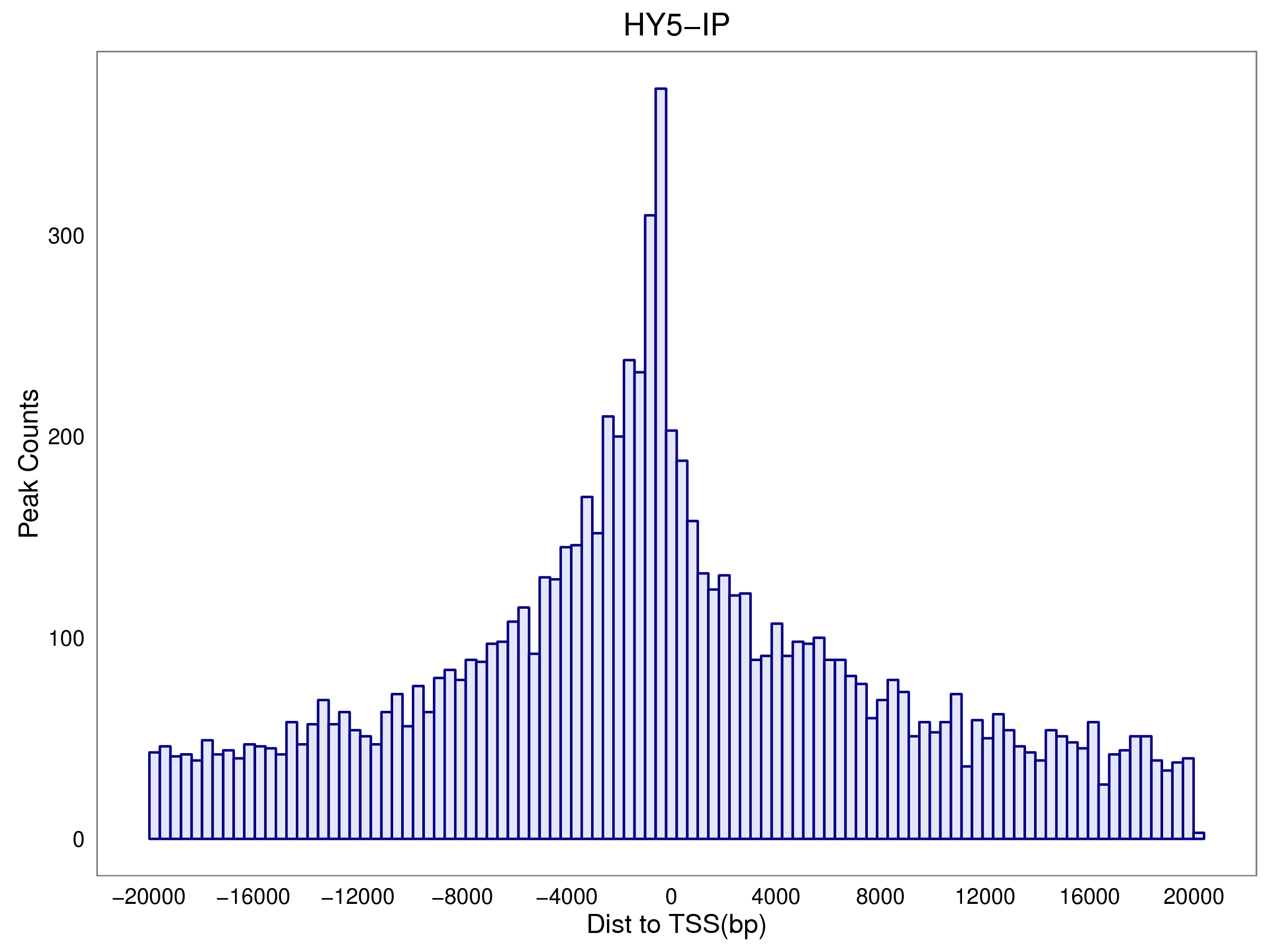 |
Fig 6-2-1 HY5-IP |
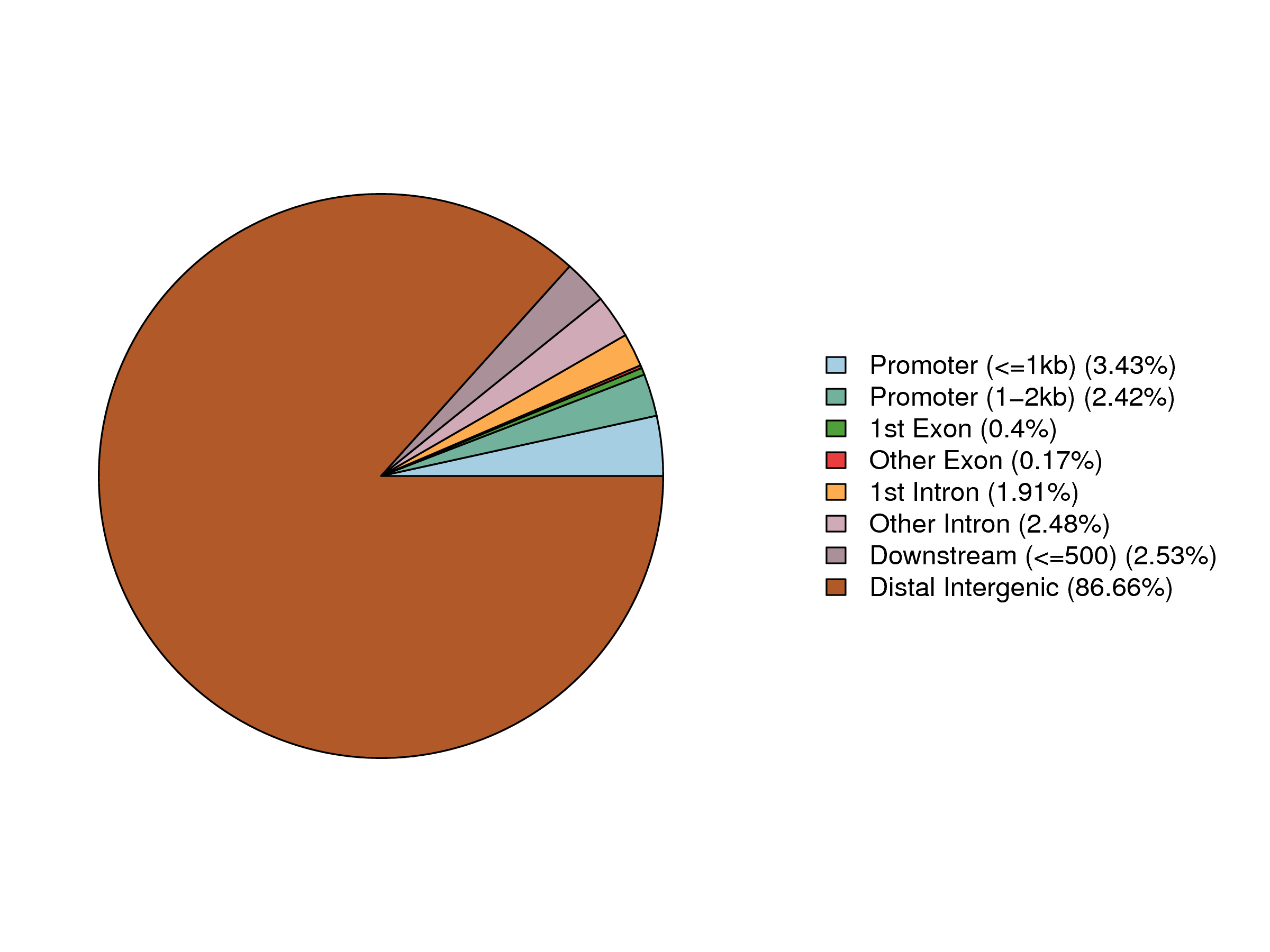 |
| Fig 6-2-2 HY5-IP |
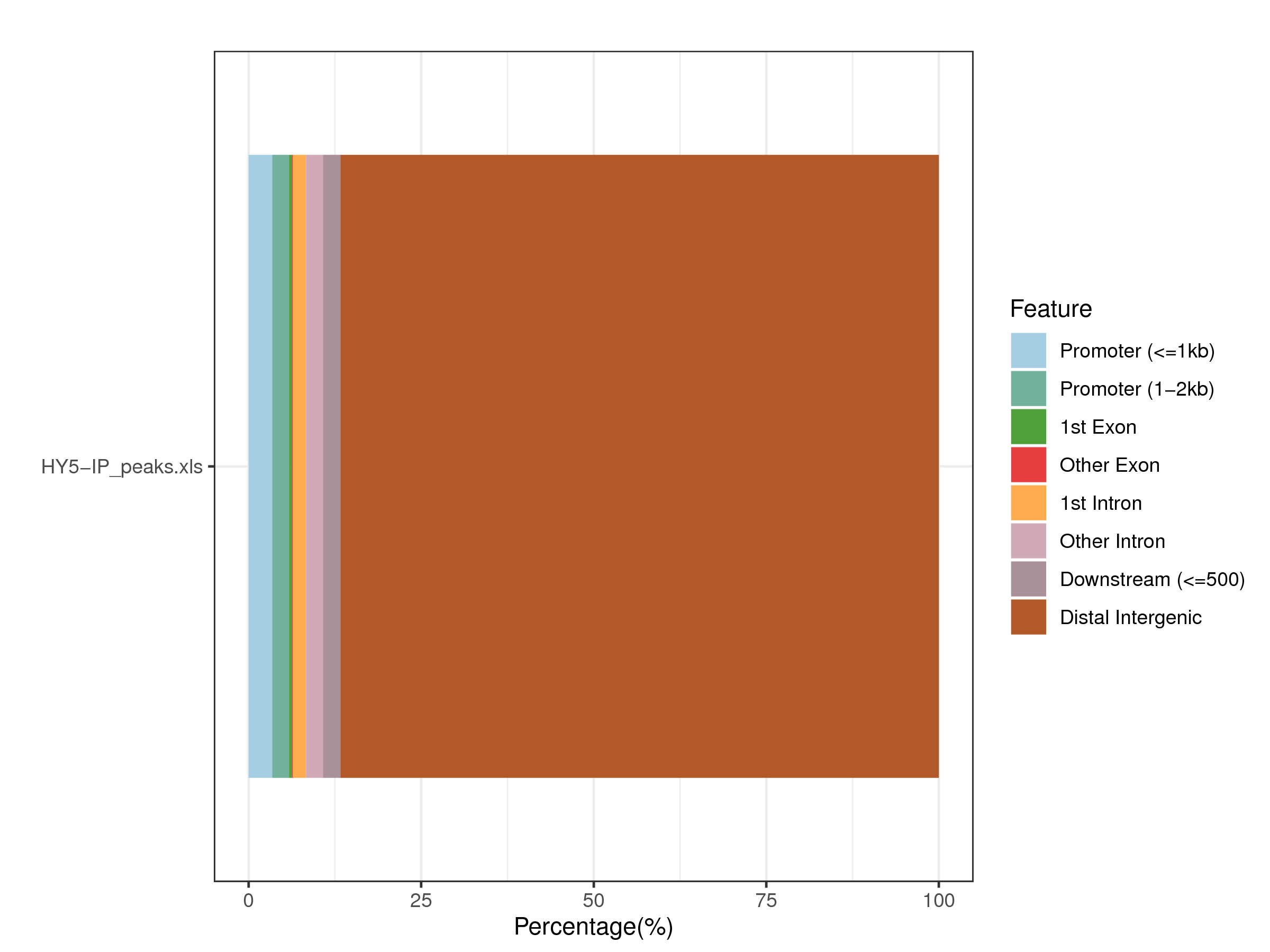 |
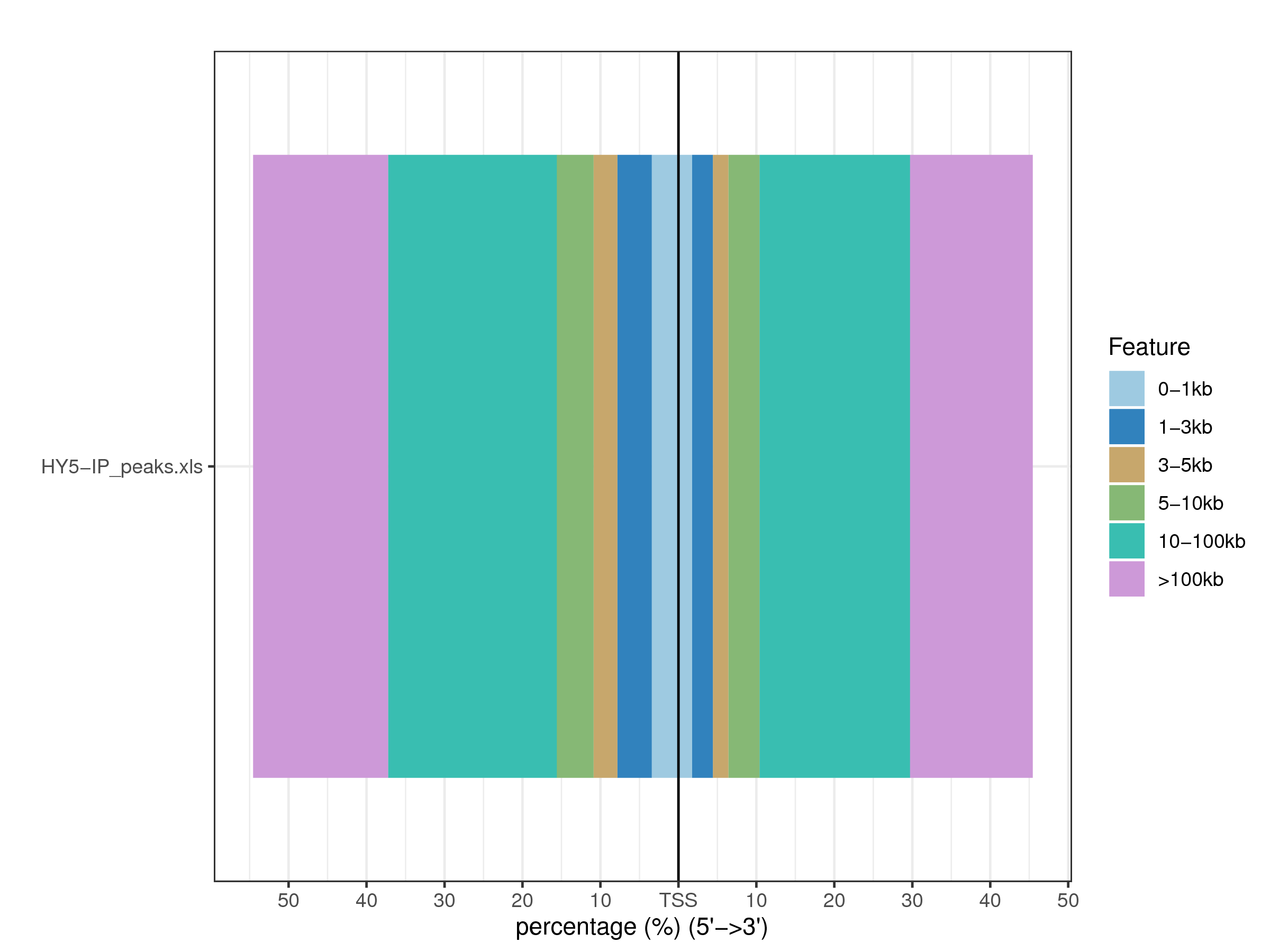 |
|
| Fig 6-2-3 各样本peak在基因功能元件上的分布比例图 | Fig 6-2-4 各样本peak相对TSS距离分布比例图 | |
6.3 Peak在染色体上的分布
 |
| Fig 6-3-1 HY5-IP |
6.4 peak相关基因 GO富集分析
Gene Ontology(简称GO)是一个国际标准化的基因功能分类体系,提供了一套动态更新的标准词汇表(controlled vocabulary)来全面描述生物体中基因和基因产物的属性。GO总共有三个ontology(本体),分别描述基因的分子功能(molecular function)、细胞组分(cellular component)、参与的生物过程(biological process)。GO的基本单位是term(词条、节点),每个term都对应一个属性。 GO功能分析一方面给出基因的GO功能分类注释;另一方面给出基因的GO功能显著性富集分析。 首先,我们将基因向GO数据库(http://www.geneontology.org/)的各term映射,并计算每个term的基因数,从而得到具有某个GO功能的基因列表及基因数目统计。然后应用超几何检验,找出与整个基因组背景相比,在基因中显著富集的GO条目。
| 基因集 | 细胞组分 | 分子功能 | 生物学过程 | GO 分类表 |
|---|---|---|---|---|
| HY5-IP | HY5-IP.C.html | HY5-IP.F.html | HY5-IP.P.html | HY5-IP.Level2.xls |
GO富集圈图:(第一圈:富集前20的GOterm,圈外为基因数目的坐标尺。不同的颜色代表不同的Ontology; 第二圈:背景基因中该GOterm的数目以及Q值。基因越多条形越长,Q值越小颜色越红; 第三圈:该GOterm差异基因数量 第四圈:各GOterm的RichFactor值(该GOterm中差异数量除以所有数量),背景网格线,每一格代表0.1)
- HY5-IP 富集圈图
Fig 6-4-1 GO 富集圈图
GO 富集分类柱状图:(横坐标为二级GOterm,纵坐标为该term里的基因数量,不同颜色表色不同类型GOterm)
- HY5-IP
Fig 6-4-2 GO富集分类柱状图
GO富集气泡图:(利用Q值最小的前20个GOterm来作图,纵坐标为GOterm,横坐标为富集因子(该GOterm中差异数量除以所有数量),大小表示数量多少,颜色越红Q值越小)");
- HY5-IP.C 气泡图
- HY5-IP.F 气泡图
- HY5-IP.P 气泡图
Fig 6-4-3 GO富集气泡图
GO富集条形图:(利用Q值最小的前20个GOterm来作图,纵坐标为GOterm,横坐标为该GOterm数目占所有差异数目的百分比,颜色越深Q值越小,柱子上的数值为该GOterm数量及Q值");
- HY5-IP.C 富集柱形图
- HY5-IP.F 富集柱形图
- HY5-IP.P 富集柱形图
Fig 6-4-4 GO富集条形图
6.5 peak相关基因 KO富集分析
在生物体内,不同基因相互协调行使其生物学,基于Pathway的分析有助于更进一步了解基因的生物学功能。KEGG是有关Pathway的主要公共数据库。 Pathway显著性富集分析以KEGG Pathway为单位,应用超几何检验,找出与整个基因组背景相比,在基因中显著性富集的Pathway。通过Pathway显著性富集能确定基因参与的最主要生化代谢途径和信号转导途径。
| 基因集 | Pathway 富集结果 | Pathway 注释表 |
|---|---|---|
| HY5-IP | HY5-IP.htm | HY5-IP.path.xls |
KO富集圈图:(第一圈:富集前20的pathway,圈外为基因数目的坐标尺。不同的颜色代表不同的A class; 第二圈:背景基因中该pathway的数目以及Q值。基因越多条形越长,Q值越小颜色越红; 第三圈:该pathway差异基因数量 第四圈:各pathway的RichFactor值(该pathway中差异数量除以所有数量),背景网格线,每一格代表0.1)
- HY5-IP 富集圈图
Fig 6-5-1 KO 富集圈图
KO富集气泡图:(利用Q值最小的前20个pathway来作图,纵坐标为pathway,横坐标为富集因子(该pathway中差异数量除以所有数量),大小表示数量多少,颜色越红Q值越小)");
- HY5-IP
Fig 6-5-2 KO富集气泡图
KO富集条形图:(利用Q值最小的前20个pathway来作图,纵坐标为pathway,横坐标为该pathway数目占所有差异数目的百分比,颜色越深Q值越小,柱子上的数值为该pathway数量及Q值");
- HY5-IP 富集柱形图
Fig 6-5-3 KO富集条形图
6.6 Peak以及周边基因结构的可视化
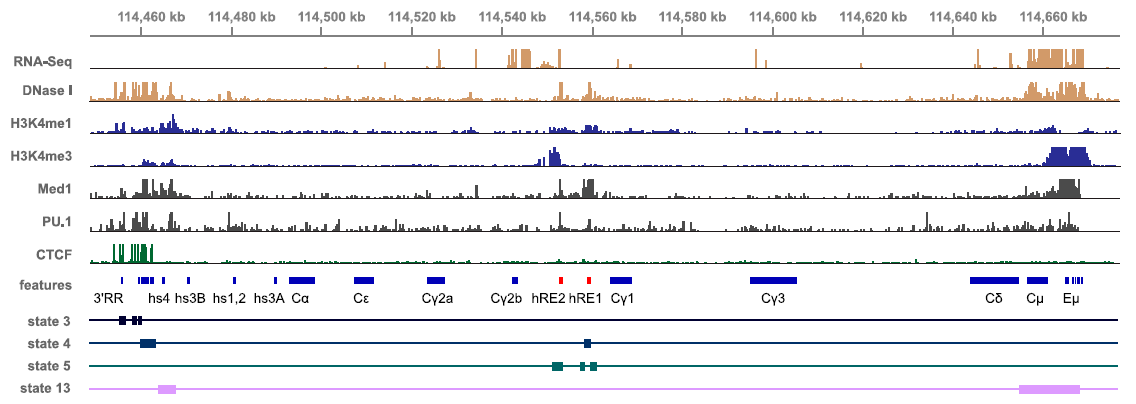 |
| Fig 6-6-1 基因结构可视化 |
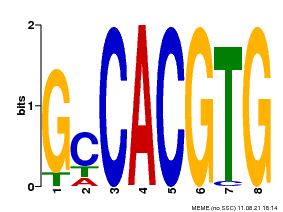
7 Motif分析
7.1 motif检测
- 样本HY5-IP meme-chip网页版结果报告: /5.motif/HY5-IP/meme-chip/meme-chip.html
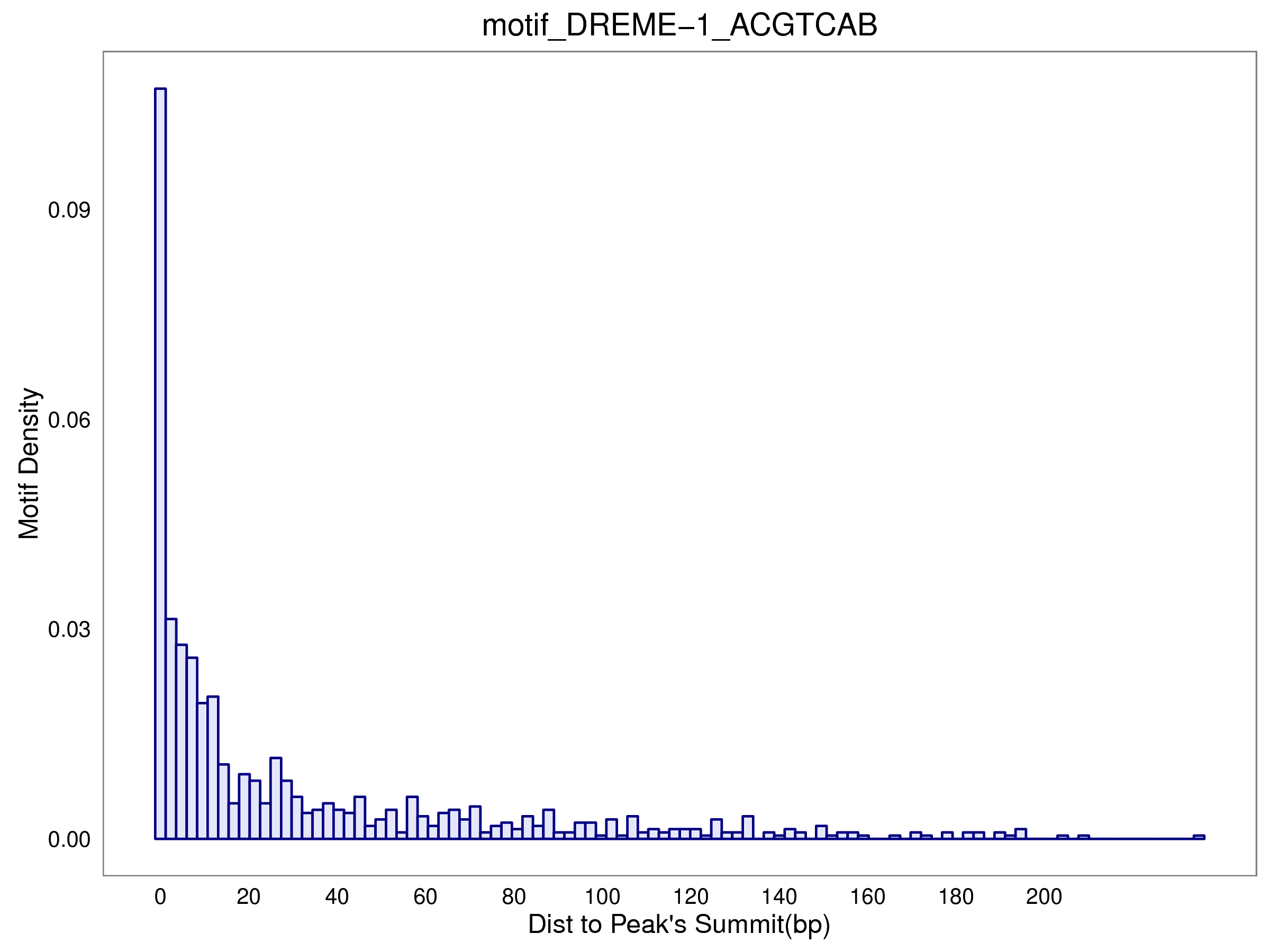
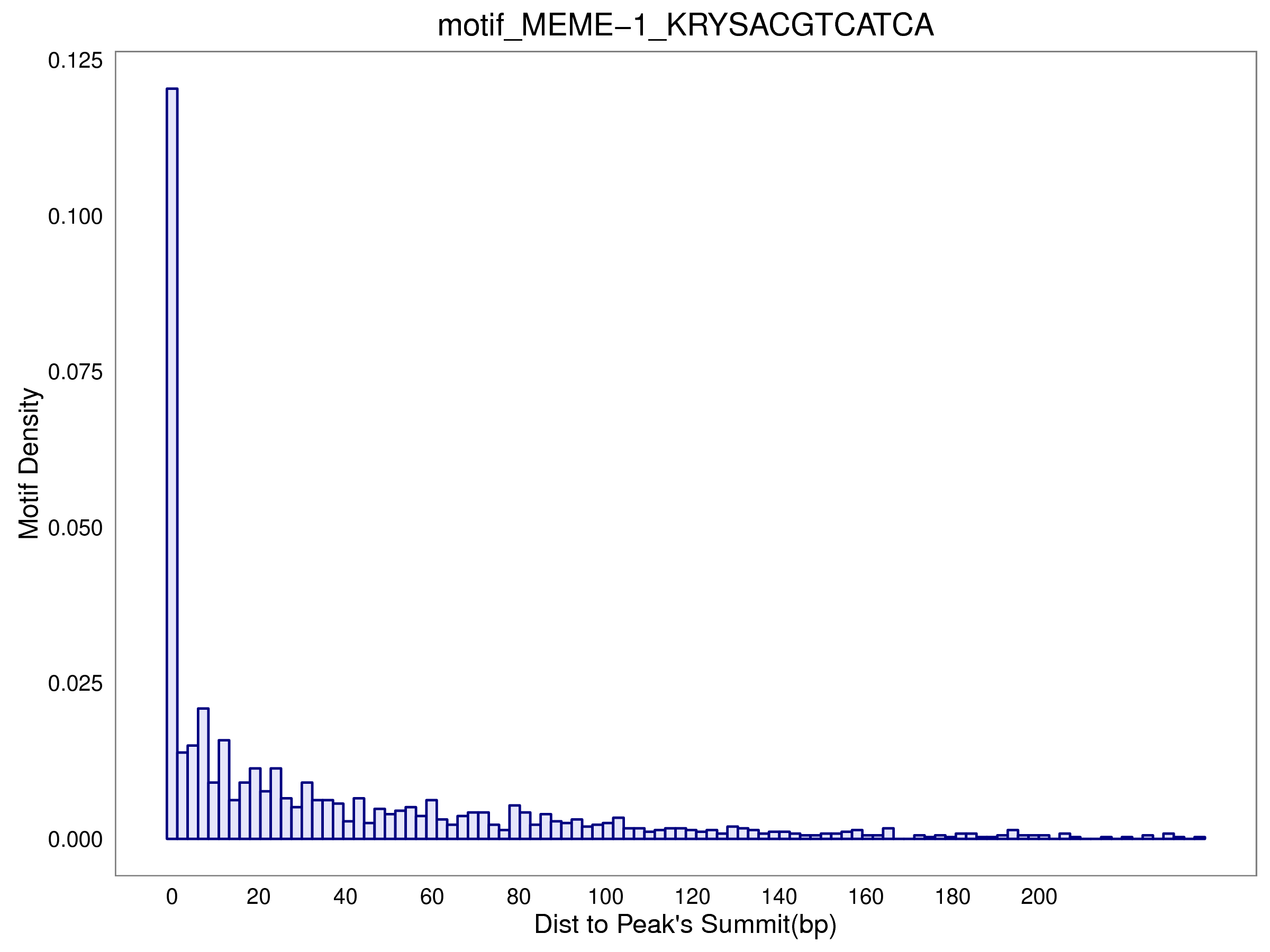
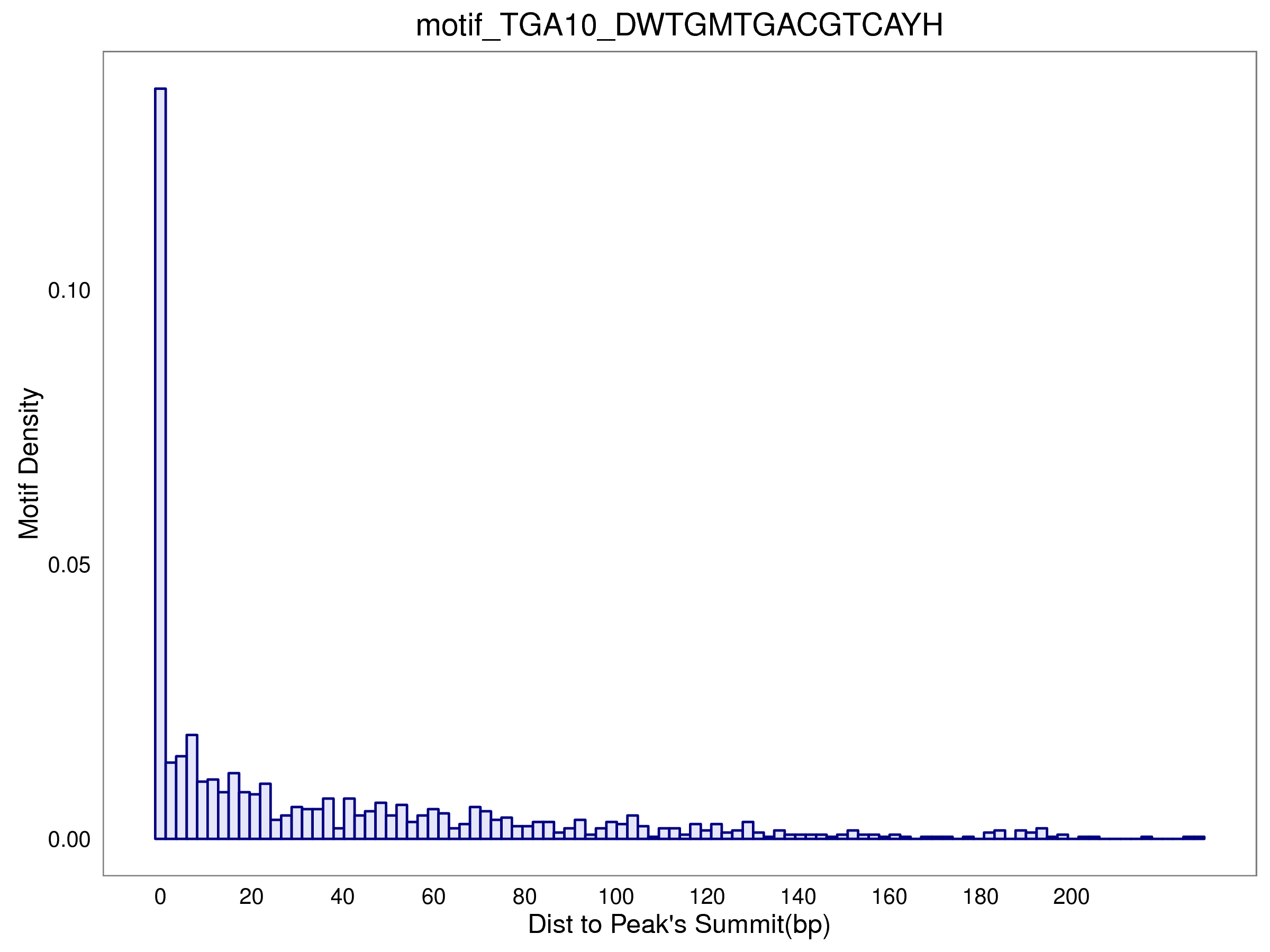
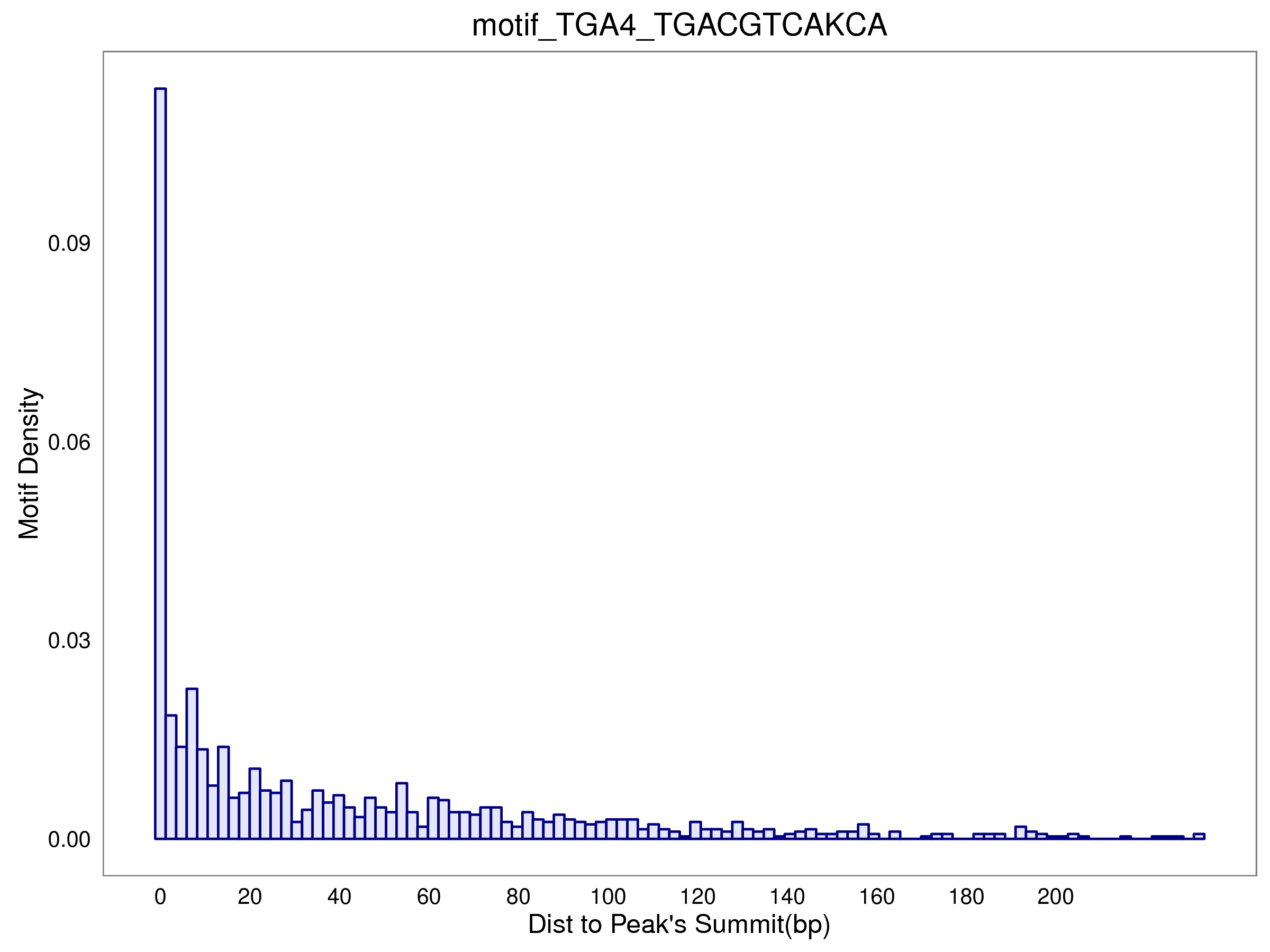
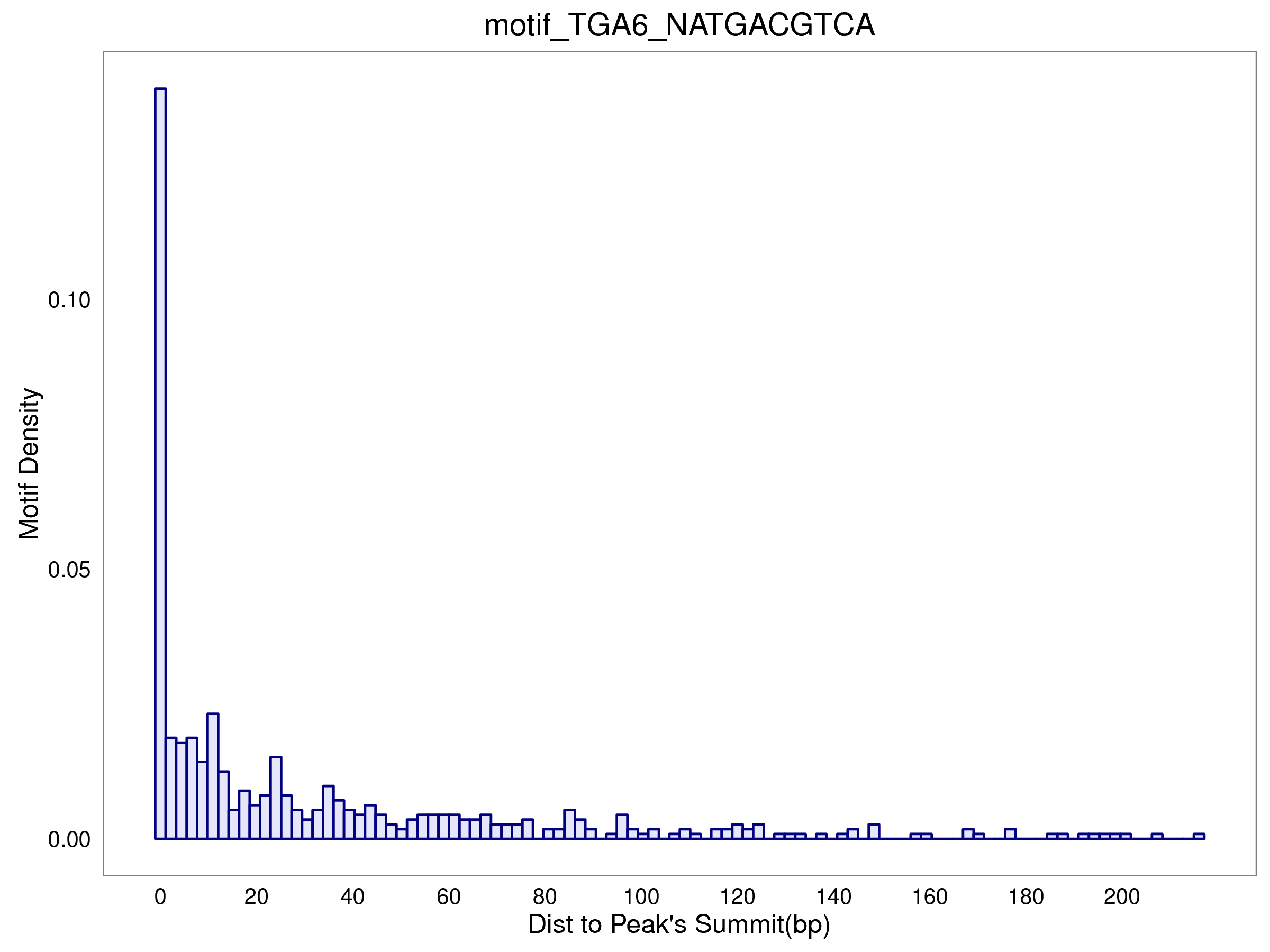
7.2 各motif到peak顶点(submit)的距离统计
- 样本HY5-IP fimo motif位置统计表: /5.motif/HY5-IP/fimo/HY5-IP.motif_anno.xls
7.3 motif注释
- 样本HY5-IP meme tommom网页版结果报告: /5.motif/HY5-IP/meme-chip/meme_tomtom_out/tomtom.html
- 样本HY5-IP dreme tommom网页版结果报告: /5.motif/HY5-IP/meme-chip/dreme_tomtom_out/tomtom.html
8 目录结构
upload ├── 1.data_access 过滤统计目录 │ ├── all.read.stat.xls 过滤信息统计表 │ ├── all.data.stat.xls 过滤前后碱基信息统计表 │ ├── all.count.{png,pdf} 样本过滤分析频数图 │ ├── all.fill.{png,pdf} 样本过滤分析比例图 │ ├── *.old.png 各样品过滤前各项统计图 │ └── *.new.png 各样品过滤后各项统计图 ├── 2.alignment 比对统计结果 │ ├── sample.alignstat.xls 各样品比对参考基因组统计结果 │ ├── *.cov.{png,pdf} 测序深度累积分布统计图 │ ├── *.tss_tes.{png,pdf} 测序深度分布图 │ ├── *.saturation.{png,pdf} 测序饱和度分布图 │ └── *.chr.{png,svg} reads在染色体上的分布图 ├── 3.peak_analysis Peak分析结果 │ ├── sample.peakstat.xls 各样本 peak 数目统计表 │ ├── *.peak.len.{png,pdf} Peak长度分布 │ ├── *.peak.dep.{png,pdf} Peak深度分布 │ ├── *.peak.fe.{png,pdf} Peak富集倍数分布 │ └── *.peak.pv.{png,pdf} Peak置信程度分布 ├── 4.peak_annotation Peak注释 │ ├── all.annostat.xls peak在基因功能元件上的分布统计表 │ ├── all.peakanno.bar.{png,pdf} peak在基因功能元件上的分布饼图 │ ├── all.peakanno.distToTSS.{png,pdf} peak相对TSS距离分布比例图 │ ├── *.final.anno.xls peak相关基因注释表 │ ├── *.peakanno.bar.{png,pdf} peak在基因功能元件上的分布比例图 │ ├── *.peakanno.pie.{png,pdf} peak在基因功能元件上的分布饼状图 │ ├── *.peakanno.cov.{png.pdf} peak在染色体上的分布图 │ ├── *.distToTss.{png,pdf} peak_summit相对基因位置分布图 │ └── enrich 富集结果目录 ├── 5.motif Motif分析结果 │ ├── meme-chip 样本meme-chip结果目录 │ │ └── meme-chip.html 样本meme-chip网页报告 │ └── fimo 样本fimo结果目录 │ ├── fimo.html 样本fimo网页报告 │ ├── *.motif.dist.png 样本motif距离peakSummit分布图 │ └── *.motif_anno.xls 样本motif基因注释表 ├── src 结果报告内容 │ ├── content.html 结果报告内容 │ ├── css 结题报告css脚本 │ ├── js 结题报告js脚本 │ ├── doc 结题报告说明文档 │ └── image 结题报告图片 └── index.html 网页版结题报告
9 参考文献
- [1] Langmead B, Salzberg S L. Fast gapped-read alignment with Bowtie 2[J]. Nature methods, 2012, 9(4): 357. 返回
- [2] Fidel Ramírez, Ryan D P , Björn Grüning, et al. Deeptools2: A next generation web server for deep-sequencing data analysis[J]. Nucleic Acids Research, 2016, 44(Web Server issue):gkw257. 返回
- [3] Zhang Y, Liu T, Meyer CA, et al. Model-based Analysis of ChIP-Seq (MACS). Genome Biology, 2008, 9: R137 返回
- [4] Yu G, Wang L G, He Q Y. ChIPseeker: an R/Bioconductor package for ChIP peak annotation, comparison and visualization[J]. Bioinformatics, 2015, 31(14): 2382-2383. 返回
- [5] MEME suit : http://meme-suite.org/ 返回
10 附录
10.1 分析方法英文文档
分析方法文档(英文):DAP-seq_method.pdf
10.2 结果文件查看
*.xls,*.txt :结果数据表格文件,文件以制表符(Tab)分隔。unix/Linux/Mac用户使用 less 或 more 命令查看;windows用户使用高级文本编辑器Notepad++ 等查看,也可以用Microsoft Excel打开。
*.png:结果图像文件,位图,无损压缩。
*.pdf:结果图像文件,矢量图,可以放大和缩小而不失真,方便用户查看和编辑处理,可使用Adobe Illustrator进行图片编辑,用于文章发表等。
10.3 文章引用与致谢
如果您的研究课题使用了基迪奥的测序和分析服务,我们期望您在论文发表时,在Method部分或Acknowledgements部分引用或提及基迪奥公司。以下语句可供参考:
- Method部分:The cDNA/DNA/Small RNA libraries were sequenced on the Illumina sequencing platform by Genedenovo Biotechnology Co., Ltd (Guangzhou, China).
- Acknowledgements部分:We are grateful to/thank Guangzhou Genedenovo Biotechnology Co., Ltd for assisting in sequencing and/or bioinformatics analysis.